この記事は、インテル® デベロッパー・ゾーンに公開されている「Enable DNA Storage on Heterogeneous Architectures with oneAPI」の日本語参考訳です。
この記事の PDF 版はこちらからご利用になれます。
OneJoin はデータ並列 C++ (DPC++) を使用して、異なる計算システム間で「編集類似性結合」アルゴリズムを実行し、大規模な DNA 配列を読み取る際の重要な問題を解決します。

課題
データ生成の急速な成長と、それらのデータを保存するための企業やクラウド・プロバイダーのニーズから、DNA (デオキシリボ核酸) など新しい記憶媒体を利用した研究が進められています。年率 60%1 でのデータの成長は、従来の磁気媒体やシリコン媒体の集積度の年率 10 ~ 30%2 の増加 (クライダーの法則) を上回っています。従来の媒体と比較して、次の特性を備えた DNA はアーカイブや低温保存に適しています (図 1)。
- 非常に高密度: LTO-8 磁気テープ・カートリッジの容量が 30TB であるのに対し、1 グラムの DNA には理論的に 455 エクサバイトのデータを保存することができます。
- 信頼性: 現代において古代の生物学的標本から DNA を抽出できるように、DNA は低温で乾燥した環境下で数千年から数十万年の間持続することが明らかになっています。
- 技術の変化がない:DNA の集積度は固定されているため、ほかのストレージ技術のように時間の経過とともに技術が進歩してアーキテクチャーが変化することはありません。
- 低コストで「人工複製」が可能。
DNA 分子は、アデニン (A)、シトシン (C)、グアニン (G)、チミン (T) の 4 つのヌクレオチドが対になって配列された二重らせん構造です。データ保存に使用される DNA は、これらのヌクレオチドの一本鎖配列であり、オリゴヌクレオチド (オリゴ) と呼ばれます。
デジタル記憶媒体として DNA を使用するには、エンコード・アルゴリズムを使用してデジタルデータをオリゴ配列にマッピングする必要があります。3 エンコードしたオリゴヌクレオチド配列は、化学的プロセスを経て DNA の合成に使用されます。合成には限界があるため、各 DNA 鎖は数百ヌクレオチド以上の長さにすることはできません。そのため、デジタルデータは何百万ものオリゴを使って保存されます。
オリゴに格納されたデータは、次世代シーケンサー (NGS) を用いて DNA 分子をシークエンシングすることで読み出されます。NGS は、DNA 鎖を複製して数百回、数千回読み取ること (ハイカバレッジ) で DNA を読み取ります。しかし、シークエンシングにはエラーが伴い、繰り返し読み出されたものが必ずしも元のオリゴと完全に一致するとは限りません。そのため、DNA に格納されたデータをデコードして元のオリゴを特定するには、多数のリードから類似するシーケンスをアライメントして (英語) 類似するリードから元のオリゴを推論するリード・コンセンサスが実行されます。推論されたオリゴは、元のデータを復元するためデコーダーに渡されます。リード・コンセンサスは、小さなデータセットを扱う場合でも計算量の多いプロセスです。大量の DNA や何億ものリードを扱う場合には、大きな課題となります。

図 1. 新しい DNA ストレージ技術は集積度が高い大規模なコールド・データ・ストレージを提供
ソリューション
Raja Appuswamy 博士は、Eurecom (英語) のデータサイエンス学部の助教です (図 2)。Eurecom は、パートナー 6 団体により設立されたコンソーシアムであり、EU の資金援助を受けた OligoArchive プロジェクト (英語) の一部です。Appuswamy 博士は、企業の OLTP、データ・ウェアハウス、計算ゲノミクスなどのデータ集約型科学領域向けの高性能データ管理システムの構築方法を研究しています。これには、データベース、バイオインフォマティクス、ストレージシステム、クラウド・コンピューティング、ハイパフォーマンス・コンピューティングなど、いくつかの分野での作業が必要となります。

図 2. Raja Appuswamy 博士と研究者の Eugenio Marinelli
Appuswamy 博士は、次のように述べています。
「私の研究における主な技術的課題は、最新のハードウェアを効果的に利用してデータを確実に保存し、効率良く処理できるスケーラブルなアルゴリズムとシステムを開発することです。」
OligoArchive プロジェクトでは、構造化されたデータベースを DNA に保存して検索する効率良いエンコード / デコード・アルゴリズムに取り組んでいます。
Appuswamy 博士は、次のように説明しています。
「今日の典型的な NGS の実行では、元のオリゴを特定するためアラインメントが必要な数億から数十億のリードを簡単に生成することができます。しかし、従来のアルゴリズムでは大規模なデータセットにスケールできません。これは、リードを比較する距離測定基準 (編集距離4 (英語)) が難解であることと、今日のサーバーで利用可能な大規模なヘテロジニアス並列性を利用できないことが原因です。」
これらの課題に対応するため、Appuswamy 博士は OneOligo プロジェクト (英語) を提案しました。
Appuswamy 博士は、次のようにコメントしています。
「OneOligo では、DNA ストレージのリード・コンセンサス問題を解決するため、データ管理 (変形性の低い埋め込みを利用した編集類似性結合 (英語) 処理) と oneAPI (英語) を利用したクロスアーキテクチャー・データ並列化を組み合わせています。」
OneOligo プロジェクトは 2 つのフェーズからなります。第 1 フェーズでは、5 カ月かけて OneJoin を開発しました。OneJoin は、DPC++ の移植性の高い並列処理を利用した、データ並列「編集類似性結合」アルゴリズムです。
Appuswamy 博士は、次のように述べています。
「OneJoin の完全に機能する DPC++ 実装が完成しました。そして、インテル® CPU、インテル® DevCloud 上の第 9 世代インテル® HD グラフィックス、さらには Codeplay の LLVM バックエンドを使用した NVIDIA* GeForce* RTX 2080 GPU など、さまざまなプロセッサー上でテストしました。我々の評価では、OneJoin が特定の重要なタスクを 100 倍以上高速化できることが実証されました (図 3)。最先端の結合実装と比較して全体では 20 倍の高速化を実現し、DNA リード・コンセンサス問題のスケーラブルなビルディング・ブロックとなりました。」
プロジェクトの第 2 フェーズでは、OneJoin をベースにスケーラブルなリード・クラスタリング・ソリューションを実装し、DNA ストレージのコンテキストでさまざまなパフォーマンスと精度のトレードオフを研究する予定です。
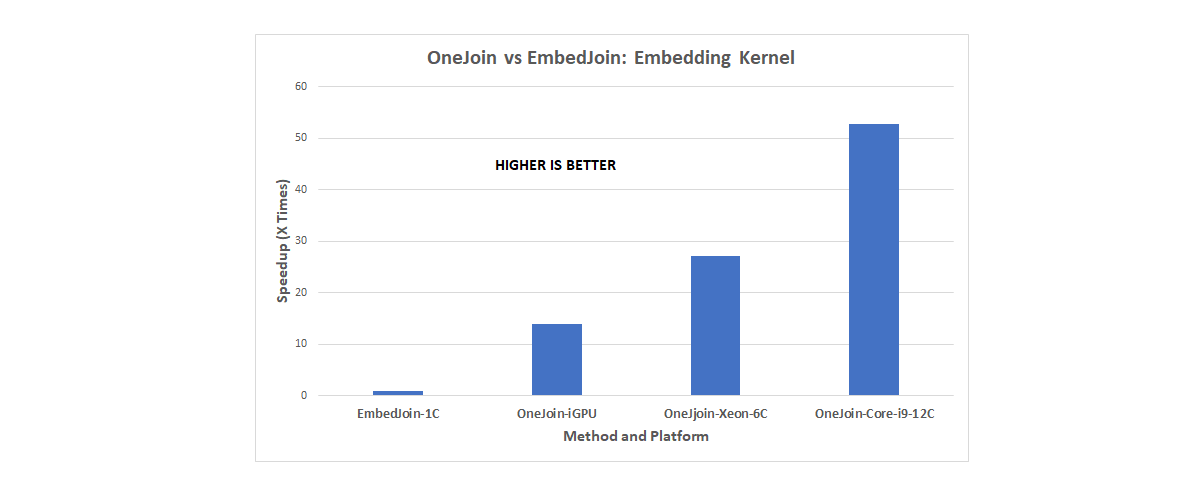 図 3. OneJoin のスピードアップ (出典: Eurecom)
図 3. OneJoin のスピードアップ (出典: Eurecom)
背景
OneOligo は、OligoArchive プロジェクト内で開発されている DNA データ保存パイプラインの計算上のボトルネックを解消する、スケーラブルなデコード・アルゴリズムです。OneOligo は、結合アルゴリズムのシングルスレッド実装から始まりました。チームは、インテル® VTune™ プロファイラーでプロファイルして、最も時間のかかるステージを特定し、それらの主要ステージのデータ並列カーネルを開発しました。これらのステップは、フォーク・ジョイン並列実行モデルの「フォーク」ステージを形成しました。
Appuswamy 博士は、次のように述べています。
「カーネルの実装には DPC++ を使用しました。これにより、単一のコードベースで複数のアーキテクチャーにカーネルをフォークすることが可能になりました。」
フォーク・ジョイン実行モデルのジョインステージはホスト上で処理されます。
Appuswamy 博士は、次のように説明しています。
「インテル® oneAPI DPC++ ライブラリー (インテル® oneDPL) とインテル® oneAPI スレッディング・ビルディング・ブロック (インテル® oneTBB) を使用して、ソートや重複排除などのさまざまなデータ構造管理タスクを並列化しました。クロスアーキテクチャーのフォーク・ジョインは、CPU と GPU を同時に利用する効果的な方法です。異なるデバイスセレクターを使用することで、これを簡単に実現できます。」
プロセス全体が 30 分以内に完了します (図 4)。
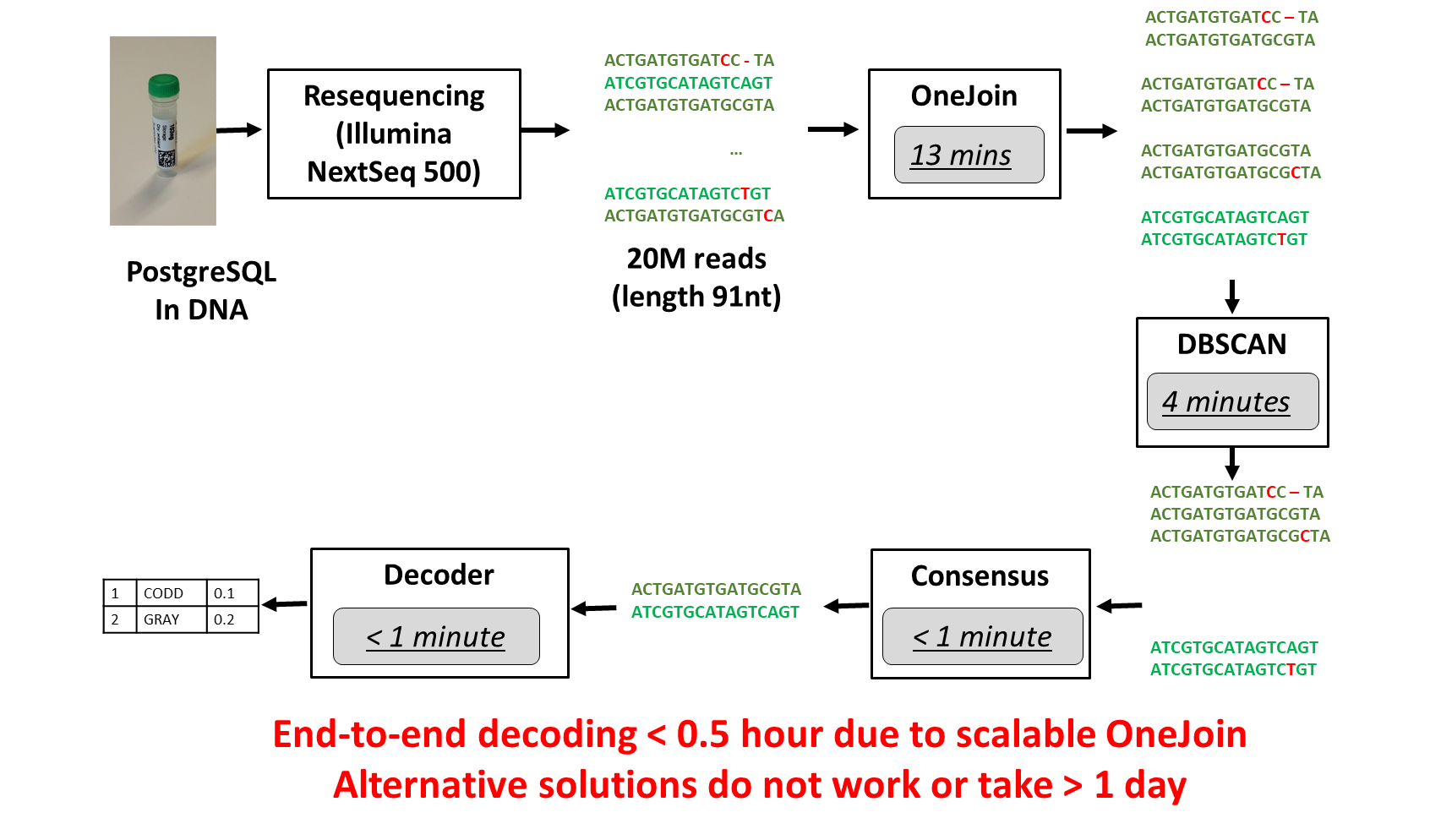
図 4. エンドツーエンドの実験プロセス (画像の出典: Raja Appuswamy 博士)
OneOligo のリード・コンセンサス手法は、OligoArchive によって DNA に保存されているデータのデコードに使用されます。Appuswamy 博士は、OneJoin のソースコードを公開し、研究論文にまとめて、OneAPI によるクロスアーキテクチャー・データ処理の研究を奨励する予定です。
Appuswamy 博士は、次のように述べています。
「我々が開発しているソリューションは、広範囲に適用可能です。例えば、OneJoin の「編集類似性結合」は、データ統合、データ・クリーニング、エンティティー認識など、データ管理領域のほかのいくつかのタスクにも使用することができます。同様に、今後開発されるリード・クラスタリング・ソリューションは、計算ゲノミクスにおける多重配列アライメントに利用することができます。」
まとめ
OligoArchive のビジョンは、DNA ストレージの採用を妨げる生物学的および計算上のボトルネックを克服することです。oneAPI を使用して、OneJoin はデコードスタックの重要なコンポーネントを実装しています。
Appuswamy 博士は、次のように締めくくっています。
「我々の知る限り、OneJoin は、さまざまなデータセットに対して拡張性と正確性を兼ね備えた唯一の編集類似性結合アルゴリズムです。スケーラブルなデータ処理において、DPC++ と oneAPI が提供するクロスアーキテクチャーの移植性に優れた並列性のメリットが明らかになりました。
oneAPI により、統一されたプログラミング・モデルを使用して、CPU や GPU (統合およびディスクリート) をターゲットとする、DNA データストレージのスケーラブルな単一ソースのデータ並列アルゴリズムを迅速に開発することができました。
我々が DPC++ を選択した理由は 2 つあります。DPC++ は、詳細なアーキテクチャー固有の最適化を行う前に、データ並列実装を素早く開発してプロトタイプをテストできます。そして、クロスアーキテクチャーとクロスプラットフォームの移植性に優れています。」
Eurecom
Raja Appuswamy 博士
使用したソフトウェアとハードウェア
Raja Appuswamy 博士は、OneJoin の開発に以下のツールや技術を使用しました。
ソフトウェア:
- インテル® oneAPI ベース・ツールキット (英語)
- インテル® Parallel Studio XE
- インテル® VTune™ プロファイラー
- インテル® oneAPI スレッディング・ビルディング・ブロック (インテル® oneTBB) (英語)
- インテル® oneAPI DPC++ ライブラリー (インテル® oneDPL) (英語)
ハードウェア:
- インテル® Xeon® E-2146G プロセッサー
- インテル® Core™ i9-10920X プロセッサー
- 第 9 世代インテル® HD グラフィックス
- インテル® デベロッパー・クラウド
リソースと推奨事項
オンラインリソース
- oneAPI (英語) に関するリソース
- oneAPI 向けインテル® DevCloud (英語)
- データ並列 C++ 書籍 (英語)
- サンプルコード (https://jamesreinders.com/dpcpp/downloads/samples20191112.tz)
- トレーニング・ビデオ (英語)
脚注
- Cold Storage in the Cloud: Trends, Challenges, and Solutions (クラウドにおけるコールドストレージ: トレンド、課題、およびソリューション) (英語)
- AIP Advances 9, 125222 (2019); https://doi.org/10.1063/1.5130404, 125222© 2019 Authors.Tape in the cloud―Technology developments and roadmaps supporting 80TB cartridge capacities (クラウド上のテープ―80TB カートリッジ容量をサポートする技術開発とロードマップ)
- Storing Digital Data into DNA: A Comparative Study of Quaternary Code Construction (DNA へのデジタルデータの保存: 4 次コード構築の比較研究) (英語)
- 指数関数的に多くの可能性の中から最適な配列を見つけなければならない編集距離の計算は、非自明な計算問題です。例えば、両方の文字列が 100 文字の場合,10^75 以上のアラインメントが考えられます。https://www.cis.upenn.edu/~cis110/12su/hw/hw06/dynprog.shtml (英語)
製品とパフォーマンス情報
1実際の性能は利用法、構成、その他の要因によって異なります。詳細については、www.Intel.com/PerformanceIndex (英語) を参照してください。


