この記事は、The Parallel Universe Magazine 49 号に掲載されている「Accelerate Single-Cell Genetics Analysis」の日本語参考訳です。原文は更新される可能性があります。原文と翻訳文の内容が異なる場合は原文を優先してください。

計測の分解能の向上は、さまざまな分野に革命をもたらしてきました。例えば、顕微鏡や望遠鏡の発明は科学に驚くほど大きなインパクトを与えました。シングルセル解析は、生物学における同様の革命の好例です。人間の身体は 40 兆個近い細胞からできています。歴史的に、これらの細胞は、ときには一度に数百万個という大きな単位で調べられてきたため、細胞間の違いをとらえることができませんでした。シングルセル解析は、細胞の個性を見るものです。新しいタイプの細胞を識別し、これらの細胞を区別するメカニズムを明らかにし、特定の病気や薬に細胞がどのように反応するかを示すことによって、細胞分化の謎が解明され始めています。この比較的新しい分野は、癌から Covid-19 関連の研究に至るまで、生物学的発見への計り知れない可能性をすでに示しています。
データ計測技術の進歩により、シングルセル・データの量は急速に増加しており、個々のデータセットのサイズも同様の速度で増加しています。このようなデータの解析には、通常、データ・サイエンス・パイプラインの実行が含まれます。パイプラインのステップは、パラメーターを変更しながら繰り返されることが多いため、ほぼリアルタイムで実行できる対話型のパイプラインが有効です。
インテル® Xeon® プロセッサー 1 基で、130 万個のマウス細胞のScRNA-seq 解析をわずか 7 分半で実現
細胞分化のさまざまな側面を研究するシングルセル解析には、多くの種類があります。シングルセル RNA-seq (scRNA-seq) 解析は、細胞間の遺伝子発現プロファイルの違いを研究します。この解析には、個々の細胞の遺伝子発現を測定する高度な技術であるシングルセル RNA シークエンシングが利用されます。
scRNA-seq 解析の典型的なワークフローは、各細胞の遺伝子発現量の行列から始まります(図 1)。データの前処理では、ノイズを除去し、データを正規化して、データセットの個々の細胞におけるすべてのヒト遺伝子の活性を取得します。このステップでは、データ収集から生じるアーティファクトを修正するため、マシンラーニングがしばしば使用されます。その後、次元縮小を行い、類似した遺伝子活性を持つ細胞をグループ化するためクラスタリングを行い、クラスターを可視化します。800,000 以上のダウンロード実績がある Scanpy (英語) は、この解析に最もよく使用されているツールキットの 1 つです。
130 万個のマウス脳細胞からなるデータセットでは、図 1 に示すパイプラインが通常、標準 Scanpy 実装(ベースライン) を使用した Google Cloud Platform* (GCP*) 上の単一 CPU インスタンス(n1-highmem-64) で 5 時間近く(英語) かかります。同じパイプラインについて、NVIDIA は、NVIDIA RAPIDS* を使用した単一の NVIDIA* A100 GPU 上で 686 秒(英語) というエンドツーエンドの実行時間を報告しています。
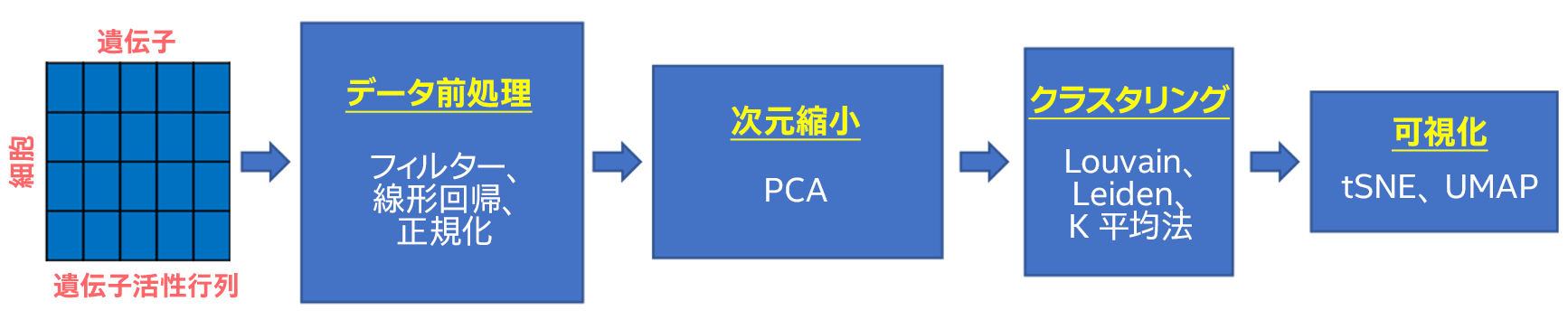
図 1. 遺伝子活性行列から始まり、異なる細胞クラスターの可視化までのシングルセルの RNA シークエンシング・データの解析手順を示すパイプライン。
インテル ラボは、インテル® oneAPI データ・アナリティクス・ライブラリー(インテル® oneDAL) チームおよび Katana Graph (英語) と協力して、より優れた並列アルゴリズムを使用してパイプラインを高速化し、アーキテクチャーに合わせてパフォーマンスをチューニングしています。この取り組みはまだ進行中ですが、表 1 と図 2 に現在のパフォーマンスとクラウド利用コストを示します。この結果は、Intel Investor Meeting 2022 (英語) で発表されました。GCP* 上の同じ単一 CPU インスタンス(n1-highmem-64) で、パイプライン全体をわずか626 秒で終了できるようになりました。第 3 世代インテル® Xeon® スケーラブル・プロセッサー(開発コード名Ice Lake) が動作する新しい n2 インスタンス・タイプでは、パフォーマンスがさらに向上しています。また、パイプラインのメモリー要件を軽減したため、ハイメモリーの n2-highmem-64 インスタンスの代わりにハイ CPU の n2-highcpu-64 インスタンスを使用できるようになりました。GCP* 上の n2-highcpu-64 のシングル・インスタンスでは、パイプライン全体がわずか 459 秒(7.65 分) で終了しています。これは、ベースラインの 5 時間と比較して約 40 倍高速です。NVIDIA* A100 GPU のパフォーマンスと比べても約 1.5 倍高速です。
高速化とメモリー要件の軽減により、クラウド・コンピューティングのコストを大幅に軽減できました(表 1)。GCP* 上の n2-highcpu-64 インスタンスのコストはわずか $0.29 です。これは、ベースライン Scanpy を実行する n1-highmem-64 の約 1/66、NVIDIA* A100 GPU の 1/2.4 です。
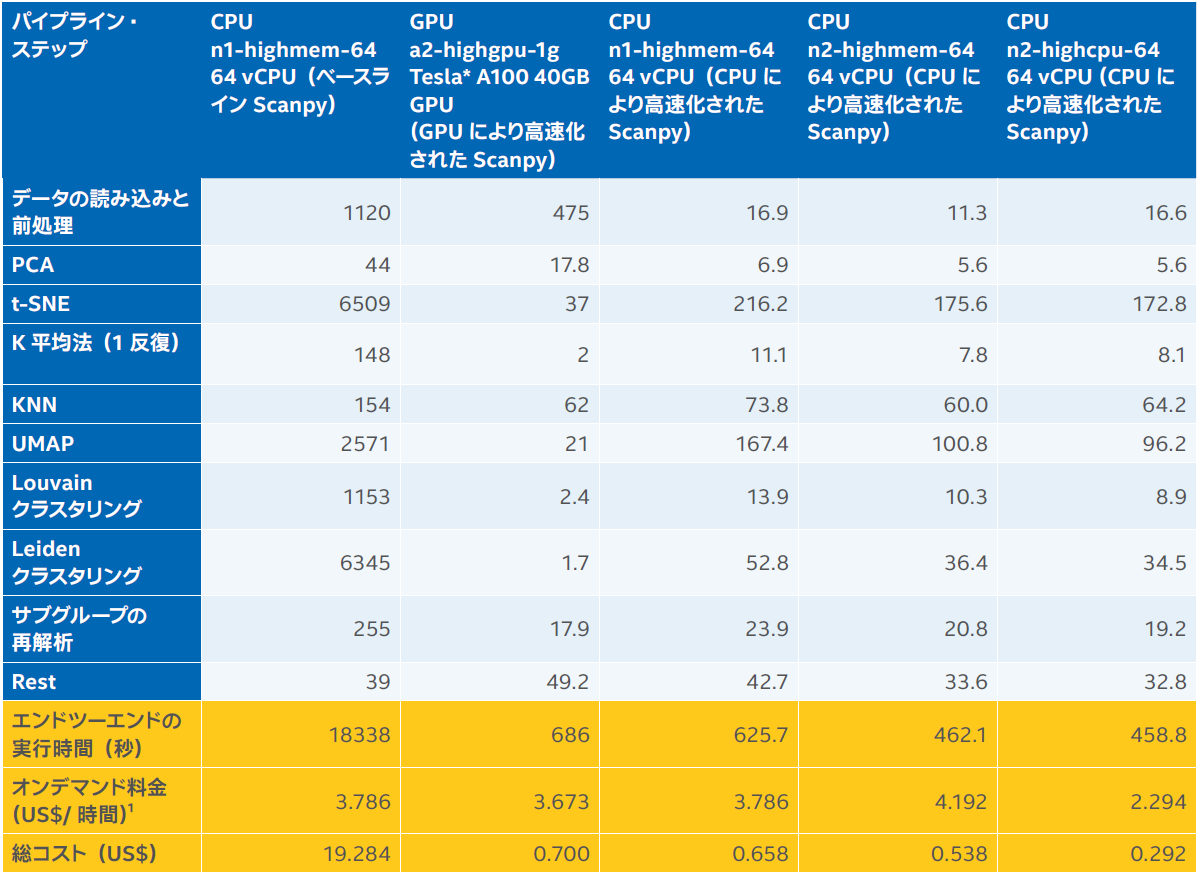
表 1. さまざまな GCP* インスタンスにおける 130 万個のマウス脳細胞の scRNA-seq 解析の実行時間とクラウドコスト。最初の 2 つのカラムは単一 CPU インスタンス(n1-highmem-64) 上のベースライン Scanpy と 単一 GPU インスタンス(a2-highgpu-1g) 上の GPU により高速化された Scanpy の公表されている(英語) 実行時間とクラウドコスト。最後の 3 つのカラムは、2 つの世代の CPU インスタンス・タイプの単一 CPU インスタンス(n1-highmem-64、n2-highmem-64、n2-highcpu-64) 上の CPU により高速化された Scanpy の測定された実行時間とクラウドコスト。
製品とパフォーマンス情報
1実際の性能は利用法、構成、その他の要因によって異なります。詳細については、www.Intel.com/PerformanceIndex (英語) を参照してください。

