この記事は、The Parallel Universe Magazine 49 号に掲載されている「Deliver Cost-Effective Genomics for Precision Medicine」の日本語参考訳です。原文は更新される可能性があります。原文と翻訳文の内容が異なる場合は原文を優先してください。

次世代シークエンシング (NGS) 技術は、全ゲノム解析および全エクソーム解析に必要なコストと時間を大幅に削減しました。NGS と効率的な二次解析により、プレシジョン・メディシン (精密医療) が臨床現場に、さらにはポイントオブケアにももたらされました。Sentieon (英語) は、ゲノム解析ソフトウェアを第 3 世代インテル® Xeon® スケーラブル・プロセッサーと第 4 世代インテル® Xeon® スケーラブル・プロセッサー (開発コード名 Sapphire Rapids) 向けに最適化しました。このソフトウェアは、マルチコアシステム上でスケーリングするように設計されており、臨床要件が迅速なターンアラウンド (例えば、救急診療で 1 人の患者のゲノムから副作用を予測する場合) であっても、高スループット (例えば、腫瘍研究室で同じ腫瘍または異なる患者の複数のサンプルを分析する場合) であっても、最高のパフォーマンスを達成できます。
Sentieon のソフトウェアは、最新のプロセッサー、特にインテル® Xeon® プロセッサー向けにベクトル化されており、独自のプログラミング言語や専用のハードウェアを使わずにハイパフォーマンスを実現するため、ベンダーへの依存を排除し、ソフトウェアの開発、展開、保守のコストを削減できます。我々は、Sentieon のパフォーマンスと精度を、NVIDIA Clara Parabricks* などの競合製品と比較し、専用ハードウェアの費用対効果と必要性を確認することにしました。
比較用の最新データは、「Benchmarking the NVIDIA Clara Parabricks Germline Pipeline on AWS (AWS* 上での NVIDIA Clara Parabricks* 生殖細胞系列パイプラインのベンチマーク) 」 (英語) で公開されています。この記事には、以下の HG001 テストのパフォーマンスとコストのデータが掲載されています。
- 全エクソーム解析 (WES) @ 50x、75x、および 100x カバレッジ
- 全ゲノム解析 (WGS) @ 30x および 50x カバレッジ
ここでは、Parabricks* と Genome Analysis Toolkit (GATK) (英語) のパフォーマンスが比較されている (図 1)、PrecisionFDA Truth Challenge (英語) の HG001 WGS 30x テストに注目します。GATK はバリアント呼び出し精度を判断する基準となっていますが、Java* で記述されているため、パフォーマンスの絶対的基準ではありません。イリノイ大学と Mayo Clinic が発表した「Sentieon DNASeq* Variant Calling Workflow Demonstrates Strong Computational Performance and Accuracy (Sentieon の DNASeq* バリアント呼び出しワークフローが強力な計算パフォーマンスと精度を実証) 」 (英語) で Sentieon が精度を落とすことなく GATK を大幅に上回ることは実証済みであるため、ここでは GATK の比較は行わず、Sentieon ソフトウェア (C++ で記述され、最新のベクトル CPU 向けに最適化されている) と Parabricks* (CUDA* で記述され、NVIDIA* GPU 向けに最適化されている) を比較します。
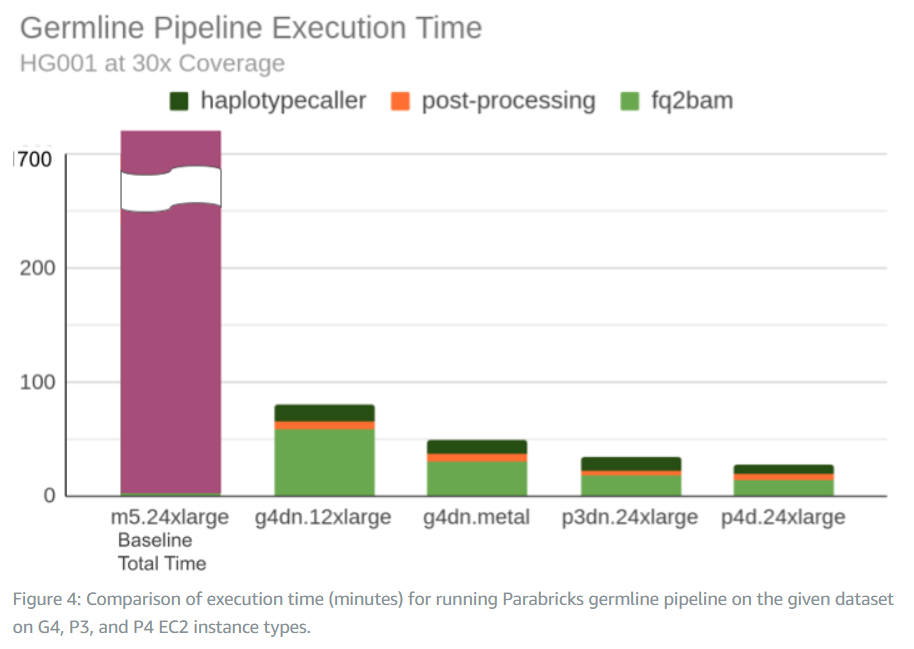
図 1. さまざまな AWS EC2* インスタンス・タイプでの GATK ベースラインに対する NVIDIA Clara Parabricks* 実行時間 (分) の比較 (出展: Benchmarking the NVIDIA Clara Parabricks Germline Pipeline on AWS (AWS* 上での NVIDIA Clara Parabricks* 生殖細胞系列パイプラインのベンチマーク)、図 4)。
Sentieon と Parabricks* のパフォーマンスをできるだけ厳密に比較するため、図 1 のベンチマークの説明とパフォーマンス・データを使用し、図 1 の haplotypecaller、後処理、fq2bam の各ステップを、バリアント呼び出しパイプラインの典型的なステージに対応付けました (表 1)。このマッピングは、Parabricks* ベンチマークに記載されている以下の記述に基づいて行いました。
「fq2bam ステップには、bwa-mem と座標ソートの一部が含まれ、後処理には座標ソートの一部、重複のマーキング、bqsr が含まれます。haplotypecaller (applybqsr) ステップは入力 bam に適用され、バリアント呼び出しステップに供給されます。」
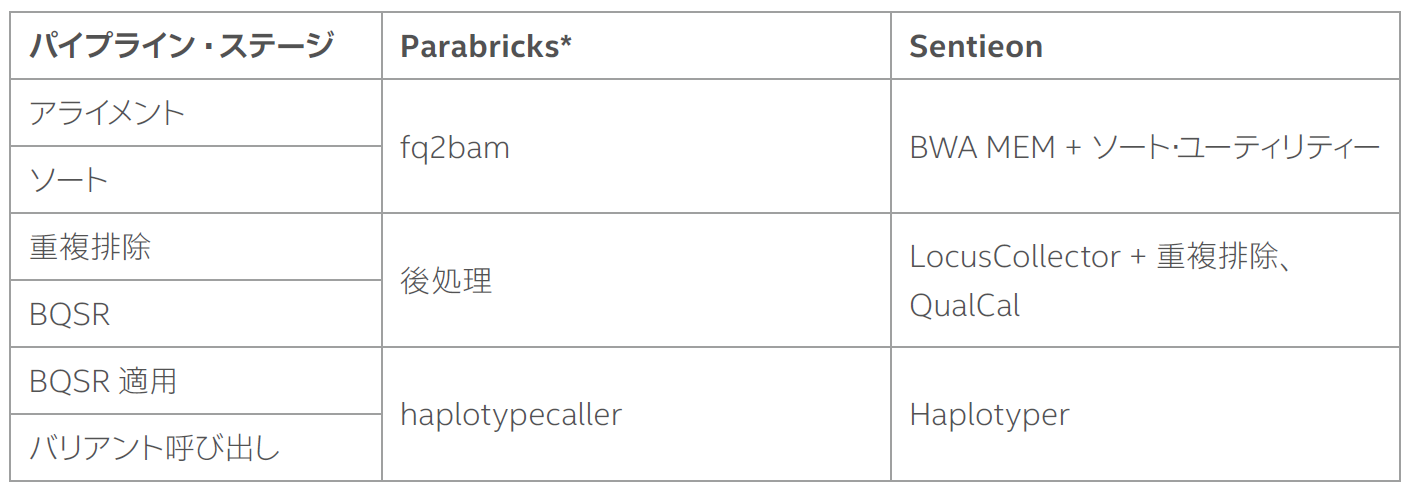
表 1. パフォーマンスの比較に使用したバリアント呼び出しパイプラインと対応する Parabricks* と Sentieon のステージ。
さまざまな計算プラットフォームにおける Sentieon と Parabricks* のパフォーマンスの比較を図 2 と表 2 に示します。プラットフォームと料金の詳細は表 3 に示します。第 3 世代インテル® Xeon® スケーラブル・プロセッサーは良好なパフォーマンスを発揮し、第 4 世代インテル® Xeon® スケーラブル・プロセッサー (開発コード名 Sapphire Rapids) は最高の総合パフォーマンスを発揮しています。しかし、パフォーマンスだけでなく、ゲノムあたりのコストと消費電力も考慮する必要があります。
インテル® Xeon® プロセッサーのゲノムあたりのコスト ($1.54) は、NVIDIA* A100 Tensor Core プロセッサー ($4.59) に比べ安価です (表 3)。第 4 世代インテル® Xeon® スケーラブル・プロセッサーの AWS EC2* 料金が同程度であれば、ゲノムあたりのコストは $1 未満になります ($2.1635/時間 * 26.8 分 = $0.97)。また、これらのベンチマークに使用された第 4 世代インテル® Xeon® スケーラブル・プロセッサーはプレリリース・ハードウェアであるため、最終製品のパフォーマンスは向上する可能性があることも特筆すべき点です。
消費電力については、c6i.metal インスタンスの 2 つのインテル® Xeon® Platinum 8352M プロセッサーが 370W を必要とするのに対し、p4d.24xlarge インスタンスの 8 つの NVIDIA* A100 Tensor Core プロセッサーは 3,200W を必要としています。Parabricks* の最高のパフォーマンスは、8.6 倍の電力と 3.0 倍のコストを必要としますが、第 3 世代インテル® Xeon® 8352M プロセッサーの 1.5 倍のパフォーマンスしか実現しません。
製品とパフォーマンス情報
1実際の性能は利用法、構成、その他の要因によって異なります。詳細については、www.Intel.com/PerformanceIndex (英語) を参照してください。

